El análisis molecular mediante PCR y electroforesis es fundamental para estudios genéticos en plantas. Este artículo titulado ¨ PCR y electroforesis de ADN extraído de hojas de tomate de árbol (Solanum betaceum) cultivado in vitro ¨presenta la aplicación de estas técnicas en ADN extraído de hojas de Solanum betaceum cultivadas in vitro, un método eficiente para obtener material vegetal estandarizado.
Tabla de Contenidos
Introducción
Los métodos de extracción de ácido desoxirribonucleico (ADN) han evolucionado desde protocolos convencionales, metodológicamente extenuantes hasta los llamados kit de extracción que son cómodos y rápidos para ser ejecutados (Rojas et al., 2014). La reacción en cadena de la polimerasa, abreviada PCR, es una técnica de laboratorio para producir (amplificar) rápidamente de millones a miles de millones de copias de un segmento específico de ADN, que luego se puede estudiar con mayor detalle. La PCR implica el uso de fragmentos cortos de ADN sintético llamados cebadores para seleccionar un segmento del genoma que se va a amplificar, y luego varias rondas de síntesis de ADN para amplificar ese segmento (Smith, 2022).
Esta técnica tiene múltiples usos y aplicaciones, la PCR puede utilizarse para detectar el ADN de una bacteria o un virus en el cuerpo de un individuo: si el patógeno está presente, es posible amplificar regiones de su ADN de una muestra de sangre o tejido, por ejemplo, Saavedra et al. (2004) mencionan que la PCR de muestras de ADN de tubérculos de papa (Solanum tuberosum L.) ha permitido el diagnóstico de Spongospora subterranea, agente causa de la enfermedad conocida como roña o sarna polvosa de la papa.
Por lo general, el objetivo de la PCR es producir suficiente ADN de la región blanco para que pueda analizarse o usarse de alguna otra manera. Por ejemplo, el ADN amplificado por PCR se puede secuenciar, visualizar por electroforesis en gel o clonar en un plásmido para otros experimentos (Reece et al., 2011).
Habitualmente, los resultados de una reacción de PCR se visualizan (se hacen visibles) al usar electroforesis en gel. La electroforesis en gel es una técnica en la que una corriente eléctrica impulsa fragmentos de ADN a través de una matriz de gel y los fragmentos de ADN se separan según su tamaño. Típicamente se incluye un estándar, o marcador de peso molecular, para que pueda determinarse el tamaño de los fragmentos en la muestra de PCR. La electroforesis es una técnica de laboratorio que se usa para separar moléculas de ADN, ARN o proteínas en función de su tamaño y carga eléctrica. Se usa una corriente eléctrica para mover las moléculas a través de un gel o de otra matriz.
Los poros del gel o la matriz actúan como un tamiz, lo cual permite que las moléculas más pequeñas se muevan más rápido que las moléculas más grandes. Para determinar el tamaño de las moléculas de una muestra, se usan estándares de tamaños conocidos que se separan en el mismo gel y luego se comparan con la muestra (NIH, 2022a; Reece et al., 2011).
El presente ensayo experimental de laboratorio tiene por finalidad la amplificación de seis muestras de ADN extraído de hojas de plántulas de tomate de árbol (Solanum betaceum L.) cultivadas in vitro y posteriormente su visualización en gel de agarosa mediante electroforesis comparando las muestras con tres reacciones con enzimas de restricción.
Objetivos
- Realizar seis reacciones en cadena de la polimerasa (PCR), una por cada muestra de ADN extraído de hojas de plántulas de tomate de árbol (Solanum betaceum L.) cultivadas in vitro.
- Realizar la visualización de las seis muestras y 3 reacciones adicionales de enzimas de restricción mediante electroforesis en gel de agarosa
- Discutir a la luz de la literatura los resultados obtenidos.
Materiales y métodos
Materiales y reactivos
- Material vegetal: seis muestras de ADN extraído de hojas de plántulas de tomate de árbol (Solanum betaceum L.) cultivadas in vitro.
- Termociclador
- Sistema de electroforesis
- Fotodocumentador
- Micropipetas
- Agua ultra pura
- TE
- DNTPs
- Buffer 10X
- Solución de MgCl2 (1.5 uM)
- Taq ADN polimerasa
- Primers (Forward and Reverse)
- Agarosa
- Buffer TBE
- Colorante de ADN: Bluejuice
- Colorante de gel
- Gradillas para tubos eppendorf
- Cinta adhesiva
- Rotuladores
Metodología
Sitio Experimental: Laboratorio de Biotecnología Vegetal de la Facultad de Ciencias Agrícolas, Universidad Central del Ecuador.
Procedimiento: Pasos de la PCR:
- Preparación de las muestras: preparar para una proyección de 7 reacciones para evitar la falta de soluciones para las muestras.
- Para preparar 6 muestras de 20 uL es necesario realizar la mezcla de los elementos que conforman la solución que contendrá el ADN a amplificarse. La tabla 1 muestra el contenido de la solución mastermix final empleando la fórmula C1V1=C2V2.
Tabla 1. Mezcla para la reacción PCR.
| Ingredientes | x1 reacción | x7 reacciones |
| Agua ultrapura | 13.8 uL | 96.6 uL |
| DNTPs | 0.4 uL | 2.8 uL |
| Buffer 10X | 2 uL | 14 uL |
| MgCl2 | 0.6 uL | 4.2 uL |
| Primer F | 0.5 uL | 3.5 uL |
| Primer R | 0.5 uL | 3.5 uL |
| Taq ADN polimerasa | 0.2 uL | 1.4 uL |
| ADN (Muestra) | 2 uL | 2 uL |
| Total | 20 uL | 128 uL |
Desnaturalización (96 °C): la reacción se calienta bastante para separar, o desnaturalizar, las cadenas de ADN. Esto proporciona los moldes de cadena sencilla para el siguiente paso.
Templado (55 – 65 °C): la reacción se enfría para que los cebadores puedan unirse a sus secuencias complementarias en el molde de ADN de cadena sencilla.
Extensión (72 °C): la temperatura de la reacción se eleva para que la Taq polimerasa extienda los cebadores y sintetice así nuevas cadenas de ADN.
Este ciclo se repite 25 – 35 veces en una reacción de PCR típica, que generalmente tarda 2 – 4 horas, según la longitud de la región de ADN que se copia.
Para la electroforesis:
- Preparar el gel al 1 % de agarosa, colocando 40 mL de buffer TBE + 0.4 g de agarosa.
- El buffer TBE se coloca en un matraz y se agita y luego se calienta en el microondas unos 20 segundos.
- Dejar enfriar la solución.
- Agregar el colorante de ADN (colorante ciber C de Invitrogen) (4 uL).
- Con cinta adhesiva se le da forma los bordes de la cámara del gel para no desbordarlo.
- Se coloca el peine para dar forma a los pocillos (10 en total).
- Se transfiere el contenido del matraz al molde y se espera solidifique.
- Se agrega 1 uL de colorante de carga para cada muestra sobre un Parafilm formando gotitas pequeñas.
- Se cargan las muestras con el colorante y se van colocando en los posillos en orden (1 uL de colorante + 5 uL de producto homogenizados), lo mismo para los productos de las enzimas de restricción y adicional se agrega un marcador de pesos moleculares, de pesos conocidos.
- Se colocan en los pocillos con la micropipeta.
- EL gel esta totalmente sumergido en TBE para movilizar las muestras por carga.
- Se tapa y se activa el sistema por al menos 1 hora.
Resultados
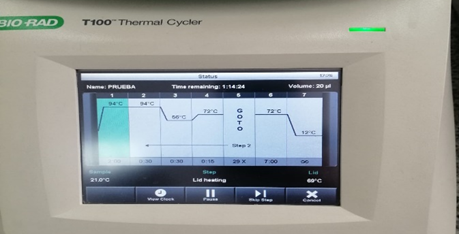
Productos de la PCR se obtuvieron 6 productos que se amplificaron de las muestras de ADN, anteriormente extraídas como se observa en la figura 1 de los anexos, la figura 2 muestra la programación del termociclador. La figura 3 muestra la cámara de electroforesis lista para su funcionamiento ya con la carga de las muestras. Se realizó la visualización de los resultados de la electroforesis en un fotodocumentador de fluorescencia donde se evidenciaron diferentes pesos moleculares entre los productos de la digestión con enzimas de restricción y el marcador de pesos moleculares como se observa en la figura 4 de los anexos.



Discusiones
De los resultados se destaca que las 6 muestras de ADN de hoja de plántulas de tomate de árbol cultivado in vitro tienen el mismo peso molecular, de entre 1100 a 1200 pb, basados en el principio de que el marcador de pesos moleculares empleado aumenta en tamaño de 100 en 100 pb. Estos resultados muestran mayor contenido de pb que los resultados publicados por Ordoñez et al. (2009) quienes obtuvieron que los pesos de las bandas encontradas variaron entre 800 a 100 pb usando la técnica AFLP.
Las bandas utilizadas para el análisis fueron las que presentaron mayor definición en el gel de acrilamida; y fueron las bandas comprendidas entre los 200 pb y los 600 pb, para Solanum betaceum. Esta variación se debe a que se puede tratar de fragmentos de ADN distintos con polimorfismos distintos. Por ejemplo, según XXXX para el gen GUS se esperarían unos 1028 pb mientras que para el gen NPTII se esperarían unas 804 pb, ambos genes presentes en Solanum betaceum (Arias, 2007; Atkinson & Gardner, 1993).
En los tres últimos pocillos en los que se colocaron los productos de las reacciones de las submuestras de ADN con enzimas de restricción se observa que la mayor fluorescencia se concentra en la zona más cercana al ánodo, es decir, fueron digeridas en trozos muy pequeños tal que terminan siendo incluso menores a 100 pb. Una enzima de restricción es una proteína aislada a partir de bacterias que cortan secuencias de ADN en sitios específicos de la secuencia, lo que produce fragmentos de ADN con una secuencia conocida en cada extremo.
El uso de enzimas de restricción es sumamente importante para determinados métodos de laboratorio, tales como la tecnología de ADN recombinante y la modificación genética (NIH, 2022b). Las enzimas de restricción son proteínas cuya función es cortar las hebras de ADN. Se las podría describir como “tijeras moleculares” que cortan ADN. Lo hacen en forma específica. Esto significa que cada enzima reconoce un sitio particular del ADN, es decir que reconoce una secuencia particular de nucleótidos.
Esa secuencia específica para cada enzima se denomina “sitio de restricción” (Real et al., 2017). Esta técnica ha resultado de valiosa ayuda en la detección de patógenos en tomate de árbol, por ejemplo, mediante RT-PCR + Elisa se ha podido confirmar la presencia de un complejo viral asociado a la enfermedad de la Virosis del tomate de árbol en los campos colombianos (Jaramillo et al., 2011).
Conclusiones
Se obtuvo mediante PCR seis amplificaciones de muestras de ADN de hojas de plántulas de tomate de árbol (Solanum betaceum) cultivadas in vitro. Mediante electroforesis se determinó que el peso molecular de las muestras fue de entre 1100 a 1200 pb. Los resultados de las digestiones con enzimas de restricción sobre el ADN muestran una senda difuminada que indica que las cadenas de ADN fueron cortadas en segmentos mucho más pequeños, la mayoría de ellos, menores a 100 pb.
Recomendaciones
Se recomendaría utilizar diferentes tipos de enzimas de restricción o de otro tipo para poder identificar mayor variabilidad en los cortes.
Se recomienda la comparación de muestras de ADN de especies de diferentes familias botánicas a fin de determinar diferencias, y de usarse mismas especies, establecer diferencias por variedades o por efecto de polimorfismos inducidos.
Referencias
Arias, A. (2007). Transformación genética de tomate de árbol (Solanum betaceum) mediada por Agrobacterium tumefaciens [(Tesis previa la obtención del título de B.S. en Biotecnología), Universidad San Francisco de Quito]. https://repositorio.usfq.edu.ec/bitstream/23000/793/1/91203.pdf
Atkinson, R. G., & Gardner, R. C. (1993). Regeneration of transgenic tamarillo plants. Plant Cell Reports 1993 12:6, 12(6), 347–351. https://doi.org/10.1007/BF00237433
Jaramillo, M., Gutiérrez, P., Cortes, J., González, E., & Marín, M. (2011). Detección de los virus AMV, CMV y PLRV en cultivos de tomate de árbol (Solanum betaceum Cav.) en Antioquia, Colombia. Rev.Fac.Nal.Agr. Medellín, 64(1), 5831–5844. http://www.scielo.org.co/pdf/rfnam/v64n1/a09v64n01.pdf
NIH. (2022a). Electroforesis. National Human Genome Research Institute. https://www.genome.gov/es/genetics-glossary/Electroforesis
NIH. (2022b). Enzima de restricción. National Human Genome Research Institute. https://www.genome.gov/es/genetics-glossary/Enzima-de-restriccion
Ordoñez, S., Torres, M., & Arahana, V. (2009). Diferenciación molecular en variedades cultivadas de tomate de árbol (Solanum betaceum), mediante la técnica molecular de AFLP. Avances, 1(1), 63–68. https://doi.org/https://doi.org/10.18272/aci.v1i1.12
Real, M., Rausell, C., & Latorre, A. (2017). Manipulación del DNA a escala genómica. In Técnicas de Ingeniería Genética (Vol. 1, pp. 443–452). Editorial Síntesis. http://www.sintesis.com
Reece, J., Urry, L., Cain, M., Wasserman, S., Minorsky, P., & Jackson, R. (2011). Amplifying DNA: The polymerase chain reaction (PCR) and its use in DNA cloning (10ma ed.). CA: Pearson.
Rojas, L., Reyes, M., Pérez-Alonso, N., Olóriz, M., Posada-Pérez, L., Ocaña, B., Portal, O., Chong-Pérez, B., & Pérez, J. (2014). Extracción in situ de ADN genómico para el análisis por PCR de regiones de interés en cuatro especies vegetales y un hongo filamentoso. Biotecnología Vegetal, 14(3), 151–154. https://revista.ibp.co.cu/index.php/BV/article/view/70
Saavedra, C., Gómez, S., & Ángel, J. (2004). Detección de secuencias específicas de ADN de Spongospora subterranea en suelo y tubérculos de papa. Revista Colombiana de Biotecnología, 6(1), 14–22.
Smith, M. (2022). Polymerase Chain Reaction (PCR). National Human Genome Research Institute. https://www.genome.gov/genetics-glossary/Polymerase-Chain-Reaction
Sugerencias
¿Te gustaría conocer más sobre cómo impulsamos la innovación y el conocimiento desde Eñengi? Visita nuestra página y descubre cómo estamos transformando el futuro a través del e-learning. ¡Haz clic aquí y únete a nuestra comunidad de cambio!







Deja una respuesta
Lo siento, debes estar conectado para publicar un comentario.