Cuantificación por espectrofotometría de una muestra de ADN extraído de plántulas de tomate de árbol (Solanum betaceum)

Introducción
En la actualidad, el estudio de la variación genética entre individuos, poblaciones y especies para explicar patrones y procesos ecológico-evolutivos y se aborda mediante marcadores moleculares, segmentos de ADN con o sin función conocida que proporcionan información sobre la variación alélica y permiten distinguir individuos (Schlötterer, 2004). Una vez obtenido el material genético, mediante extracción de ADN o ARN con técnicas como la aplicación del protocolo CETAB (Suazo et al., 2020), es importante determinar el rendimiento mediante espectrofotometría, este es el paso previo para disponer el material genético a la aplicación de técnicas moleculares aplicadas como los microsatélites, marcadores moleculares, enzimas de restricción, etc.
La ley de Beer-Lambert indica que la concentración de una molécula en solución depende de la cantidad de luz absorbida de las moléculas disueltas (Alejos et al., 2014), es decir, mediante espectrofotometría se puede determinar la concentración y la pureza de una muestra de ADN basándose en la capacidad de absorbancia de un compuesto presente en una solución a una longitud de onda determinada (Chévez, 2021).
De este modo la concentración de la muestra de ADN se calcula teniendo en cuenta el valor de absorbancia obtenido a una longitud de onda de 260 nm. Mientras que la relación de absorbancias A260/280 y A260/230 se utilizan para evaluar la pureza de las muestras. La relación A260/280 es muy estable y se considera que un ADN de pureza óptima tiene un valor entre 1.8-2.0. Un ADN de pureza aceptable debe tener al menos una relación A260/280 > 1.6 (Solano-Flórez et al., 2009), se debe considerar el factor de dilución para obtener la concentración en nanogramos/microlitro (ng/ul) (Alejos et al., 2014).
La espectrofotometría UV/Visible permite confirmar que se cuenta con cantidad suficiente de ácidos nucleicos (DNA/RNA) de calidad adecuada antes de llevar a cabo ensayos de PCR cuantitativa en tiempo real, análisis de SNPS (Polimorfismos de nucleótido único) o la secuenciación automática de muestras de DNA de plásmidos, cósmidos, productos de PCR (Marcos-Merino et al., 2019; Serrato Díaz et al., 2010).
En el presente ensayo experimental de laboratorio se ha realizado la cuantificación mediante absorción uv-visible o espectrofotometría uv-vis, de cuatro muestras de ADN extraído de hojas de plántulas de tomate de árbol generadas en medio in vitro.
Objetivos
- Cuantificar mediante espectrofotometría ADN extraído a partir de hojas de tomate de árbol generadas en medio in vitro.
- Discutir a la luz de la literatura los protocolos de cuantificación de ADN.
- Analizar los resultados de la cuantificación de seis muestras de ADN de tejidos vegetales de hojas de plántulas de tomate de árbol generadas en medio in vitro.
Materiales y métodos
Materiales y reactivos
- AND de tejidos vegetales de hojas de plántulas de tomate de árbol generadas en medio in vitro suspendido en TE en un tubo de eppendorff.
- Solución blanco de TE (Tris-EDTA).
- Micropipetas de 2 ul de volumen.
- NanoDrop One® (TermoScientific®).
- Papel toalla (Kleenex®).
Metodología
Sitio Experimental: Laboratorio de Biotecnología Vegetal de la Facultad de Ciencias Agrícolas, Universidad Central del Ecuador.
Procedimiento para cuantificar ácidos nucleicos:
- Limpiar el con una toallita Kleenex® el porta muestras ubicado en el pedestal y la cubierta del brazo.
- En la pantalla Inicio, seleccionar la opción de ácidos nucleicos, seleccionar ADNbc, ADNmc o ARN según las muestras que se requiera medir, en este caso ADN.
- Piperar 2 ul de solución blanco (Tris-EDTA) en el pedestal inferior y baje el brazo.
- Seleccione Blanco y espere hasta que se termine el proceso de estandarización del blanco.
- Levante el brazo y limpie ambos pedestales con una toallita Kleenex®, emplear papel toalla con la menor cantidad de residuos posibles, siempre usando una toallita nueva.
- Pipetee 2 ul de solución de muestra en el pedestal, baje el brazo.
- Comience la medición de la muestra con el brazo en su lugar y evitando previamente el derramamiento de muestra fuera de su área de análisis.
- Cuando haya finalizado la medición toque finalizar el experimento.
- Levante el brazo y limpie ambos pedestales con una toallita nueva.
- Repetir el procedimiento bajo el mismo blanco hasta culminar las muestras.
- Registrar los valores y analizar previo a la emisión de conclusiones.
Resultados
Se cuantificó cuatro muestras de ADN procedente de hojas de plántulas de tomate de árbol (Solanum betaceum L.) cultivadas in vitro. Se obtuvo que las seis muestras se encontraban en un rango de pureza óptimo según la relación A260/280. Los valores de concentración en ng/mL fue variable como se evidencia en la tabla de resultados (Tabla 1).
Tabla 1. Resultados de la cuantificación de 4 muestras de ADN de hojas de tomate de árbol cultivado in vitro.
| Nro. | Concentración ng/uL | A260/A280 | A260/A230 |
| Blanco TE | -0.7 | 1.48 | 0.58 |
| 1 | 1319.3 | 1.85 | 2.24 |
| 2 | 1568.2 | 1.95 | 2.36 |
| 3 | 681.2 | 1.90 | 2.38 |
| 4 | 983.3 | 1.94 | 2.37 |
Fuente: Elaboración propia.

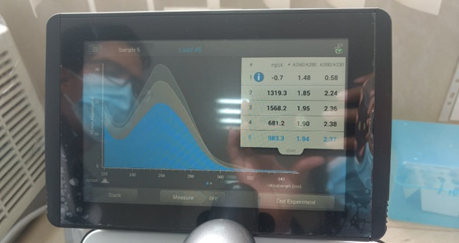
Figura 1. Sup. equipo NanoDrop One para la cuantificación de ADN; Inf. evidencia de las lecturas realizadas con el equipo.
Discusión
Los resultados arrojados en la medición de ácidos nucleicos demuestran que las 4 muestras analizadas se encuentran en un rango de absorbancia considerado como adecuado u óptimo, ya que la pureza en todos los casos fue superior a 1.8. la concentración de la muestra de ADN se calcula teniendo en cuenta el valor de absorbancia obtenido a una longitud de onda de 260 nm, la relación de absorbancias A260/280 y A260/230 se utilizan para evaluar la pureza de las muestras, la relación A260/280 es muy estable y se considera que un ADN de pureza óptima tiene un valor entre 1.8-2.0. Un ADN de pureza aceptable debe tener al menos una relación A260/280 > 1.6. Un valor A260/280 < 1.6 indica una posible contaminación por compuestos aromáticos como fenoles y proteínas. Un radio A260/280 > 2.1 podría deberse a la presencia de ARN en la muestra (Alejos et al., 2014; Benigno de los Santos-Romero et al., 2018; Sub Unidad de Soporte al Diagnóstico-Genética, 2021), en tal virtud, al observarse los resultados de la tabla 1, se evidencia que el rango medido en las 4 muestras va de 1.85 a 1.95 en la relación A260/280.
Una segunda valoración de la pureza de ácidos nucleicos es la proporción 260/230, los valores aceptados se encuentran en el rango de 2.0 a 2.2, si la relación es menor indican la presencia de contaminantes como carbohidratos o fenol (Alejos et al., 2014). Un radio menor de 1.8 se relaciona con presencia de contaminantes en la muestra. Cuanto menor sea este radio, la presencia de contaminantes en la muestra será mayor. Un radio < 1.5 estaría indicando una impureza relevante en la muestra que podría comprometer su funcionalidad, este indicador está asociado a la contaminación orgánica (Benigno de los Santos-Romero et al., 2018; Qamar et al., 2017), los valores del rango A260/230, según se muestra en la tabla 1, se consideran óptimos entre los 2.24 a 2.38, estos valores confirman que la extracción de ADN fue exitosa y no registran remanentes considerables por necesidad de azúcares, sin embargo, esta relación resulta mucho más variable que la relación A260/280 dependiendo de factores como la concentración de ADN o de la composición del tampón de resuspensión de la muestra, por lo tanto, para poder realizar una buena interpretación de los valores de absorbancia obtenidos es importante indicar la composición del tampón de resuspensión de la muestra así como el protocolo de extracción utilizado (Departamento de control de calidad del Banco Nacional de ADN Carlos III, 2020). Para evaluar la pureza de muestras de ADN en algunos procedimientos especializados como puede ser de secuenciamiento de nueva generación (NGS), específicamente en la preparación de librerías, es necesaria la detección de bajas concentraciones por espectrofotometría, ya que se trabajan diluciones de ADN de concentraciones de hasta 2ng/µL (Sub Unidad de Soporte al Diagnóstico-Genética, 2021).La extracción de reactivos, como la guanina, el fenol, y el EDTA, contribuyen a la absorbancia de 230 y 280 nm.
La concentración de ácidos nucleicos se proporciona en la unidad seleccionada (ng/uL), los cálculos se basan en una variación de la ecuación de la Ley de Beer, que utiliza el valor de absorbancia de ácidos nucleicos corregido, un valor negativo indica la inexistencia de las moléculas de interés en las muestras. Aunque los índices de pureza son indicadores importantes de la calidad de la muestra, el mejor indicador de calidad es la funcionalidad de aplicación de recuperación y purificación de interés, por ejemplo, PCR de tiempo real (Thermo Fisher Scientific Inc., 2016). Los valores de concentración estimados, según se muestra en la tabla 1, se identifica que entre la muestra 1 y 2 hay diferencias con las muestras 3 y 4, siendo estas ultimas las que tienen menor concentración de molécula, sin embargo se considera adecuado, la reducción de ácidos nucleicos en la estimación por espectrofotometría se puede dar por la naturaleza de las extracciones, el manejo de las muestras previamente, depende exclusivamente del éxito que las muestras tengan desde la extracción del ADN.
Conclusiones
Se cuantificó por espectrofotometría uv-visible 4 muestras de ADN extraído de las hojas de plántulas de Solanum betacum L. Todas las muestras obtuvieron los rangos de pureza más óptimos, las muestras analizadas corresponden al espectro de absorbancia reportado en la literatura. Las muestras presentaron alta calidad en cuanto a pureza del tipo A260/230, que confirma la inexistencia de elementos contaminantes.
Recomendaciones
Se debe blanquear con la misma solución tampón utilizada para resuspender el analito de interés, la solución blanco debe ser de igual rango de pH y fuerza iónica que la solución del analito.
Realizar un ciclo de blanqueado para valorar cómo afecta la absorbancia de la solución tampón, si dicha solución manifiesta una absorbancia importante puede ser necesario elegir una solución tampón distinta.
El pedestal y sus superficies deben permanecer limpias y acondicionadas, para evitar se contamine especialmente con microorganismos que pueden conllevar a blanqueamientos fallidos.
Homogenizar las muestras de ADN de forma previa para evitar la quema de los equipos, por lo que se sugiere someter las muestras a un vórtex.
Referencias
Alejos, L., Aragón, M. del C., & Cornejo, A. (2014). Extracción y Purificación de ADN. In A. Cornejo, A. Serrato, B. Rendón, & M. Rocha (Eds.), Herramientas moleculares aplicadas en ecología: Aspectos teóricos y prácticos (Primera Edición, Vol. 1, pp. 1–26). Secretaría de Medio Ambiente y Recursos Naturales, Instituto Nacional de Ecología y Cambio Climático y Universidad Autónoma Metropolitana – Iztapalapa. https://www.researchgate.net/publication/280731680
Benigno de los Santos-Romero, R., Pérez-León, M., Rodríguez-Magadan, H., Caballero-Sánchez, I., Montes-Yedra, J., & Gómez-Ugalde, R. (2018). Caracterización genética de la tilapia (Oreochromis niloticus) cultivada en los valles centrales de Oaxaca. Revista Mexicana de Agroecosistemas, 5(2), 128–134.
Chévez, H. (2021). Marcadores moleculares para la detección de Candidatus liberibacter causante del huanglongbing (HLB) enverdecimiento de los cítricos en el Ecuador [Universidad Agraria del Ecuador]. https://cia.uagraria.edu.ec/Archivos/CHEVEZ%20VERA%20HECTOR.pdf
Departamento de control de calidad del Banco Nacional de ADN Carlos III. (2020). Programa de control de calidad de muestras de ADN y ARN. In Departamento de control de calidad del Banco Nacional de ADN. https://cit.ligue-cancer.net/CIT_Public/images/stories/CIT/pdf/WebSite%20CIT-%20QC%20PF%20Saint-
Marcos-Merino, J., Gallego, R., & Gómez, J. (2019). Extracción de ADN con material cotidiano: desarrollo de una estrategia interdisciplinar a partir de sus fundamentos científicos. Educación Química, 30(1), 58–69. https://doi.org/10.22201/FQ.18708404E.2019.1.65732
Qamar, W., Khan, M. R., & Arafah, A. (2017). Optimization of conditions to extract high quality DNA for PCR analysis from whole blood using SDS-proteinase K method. Saudi Journal of Biological Sciences, 24(7), 1465–1469. https://doi.org/10.1016/J.SJBS.2016.09.016
Schlötterer, C. (2004). The evolution of molecular markers — just a matter of fashion? Nature Reviews Genetics , 5(1), 63–69. https://doi.org/10.1038/nrg1249
Serrato Díaz, A., Flores Rentería, L., Aportela Cortez, J., & Sierra Palacios, E. (2010). PCR: reacción en cadena de la polimerasa. In Herramientas moleculares aplicadas en ecología (1st ed., Vol. 1, pp. 53–73). Instituto de Ecología, Universidad Nacional Autónoma de México. www.biologia.
Solano-Flórez, G., Márquez-Cardona, M., & Schuler, I. (2009). Optimización de la extracción de ADN de Passiflora ligularis para el análisis por medio de marcadores moleculares. Universitas Scientiarum, 14. https://doi.org/10.11144/javeriana.sc14-1.odle
Suazo, T., Miranda, S., Rivers, E., Lacayo, M., & Tenorio, D. (2020). Evaluación de metodologías de extracción de ADN de plantas recalcitrantes. Revista Torreón Universitario, 9(24), 45–57. https://doi.org/10.5377/TORREON.V9I24.9723
Sub Unidad de Soporte al Diagnóstico-Genética. (2021). Extracción de ADN a partir de sangre periférica y médula ósea.
Thermo Fisher Scientific Inc. (2016). Espectrofotómetros micro-UV/Vis NanoDrop. Guía de usuario.
Un comentario
Deja un comentario
Lo siento, debes estar conectado para publicar un comentario.







Reblogueó esto en BIG DATA Experience, 09 de Octubre 2013, JW Lima Marriot Hotel.